
2009年内蒙古极端干旱条件下谷子的抗旱表现
近日,中国农科院作科所刁现民团队联合中科院遗传发育所赵美丞课题组在The Plant Cell上发表了题为“The osmotic stress–activated receptor-like kinase DPY1 mediates SnRK2 kinase activation and drought tolerance in Setaria”的研究论文,该研究以谷子为模式体系,揭示了DPY1(DROOPY LEAF1)在感知渗透势变化及信号向胞内转导中发挥着关键作用,为该领域研究提供了重要信息。
该团队前期在谷子中克隆了一个LRR型膜蛋白受体激酶DPY1,并发现DPY1负向调节早期油菜素内酯(BR)信号,防止其过度激活,从而维持谷子叶片坚实度与直立性(点击查看:PNAS | 我国科学家以谷子为模式揭示作物株型调控新机制)。研究人员利用LC-MS/MS筛选DPY1的互作蛋白时发现,SnRK2激酶家族成员SAPK6及其上游特异的B4家族RAF20激酶是DPY1的互作蛋白(但DPY1在体外不能直接磷酸化二者),暗示了DPY1参与了渗透胁迫信号;激酶实验表明渗透胁迫处理谷子叶片在4小时后可以显著增加DPY1的磷酸化并持续激活其激酶活性;基于TMT标记的蛋白磷酸化组分析发现DPY1的缺失会导致超过50%的渗透胁迫响应磷酸化位点失去响应(图1),其中包含了:SnRK2家族的SAPK6、MAPK级联信号成员以及质膜H+-ATPase (在拟南芥中这些蛋白被证实在早期渗透胁迫信号转导中起着关键作用)。此外,SAPK6的干旱/高渗激活依赖于DPY1(图1);遗传学及转录组分析证实SAPK6位于DPY1下游参与谷子渗透胁迫信号转导及抗旱响应,且该过程很大程度不依赖DPY1介导的BR信号。膜蛋白受体激酶在多种内外源信号的感知与转导过程中起着关键作用,但其在植物渗透胁迫信号转导中是否起着类似作用一直不清楚,该研究鉴定了一个对渗透胁迫信号转导起关键作用的膜蛋白受体激酶,并将核心响应激酶SnRK2的激活关联到细胞膜。
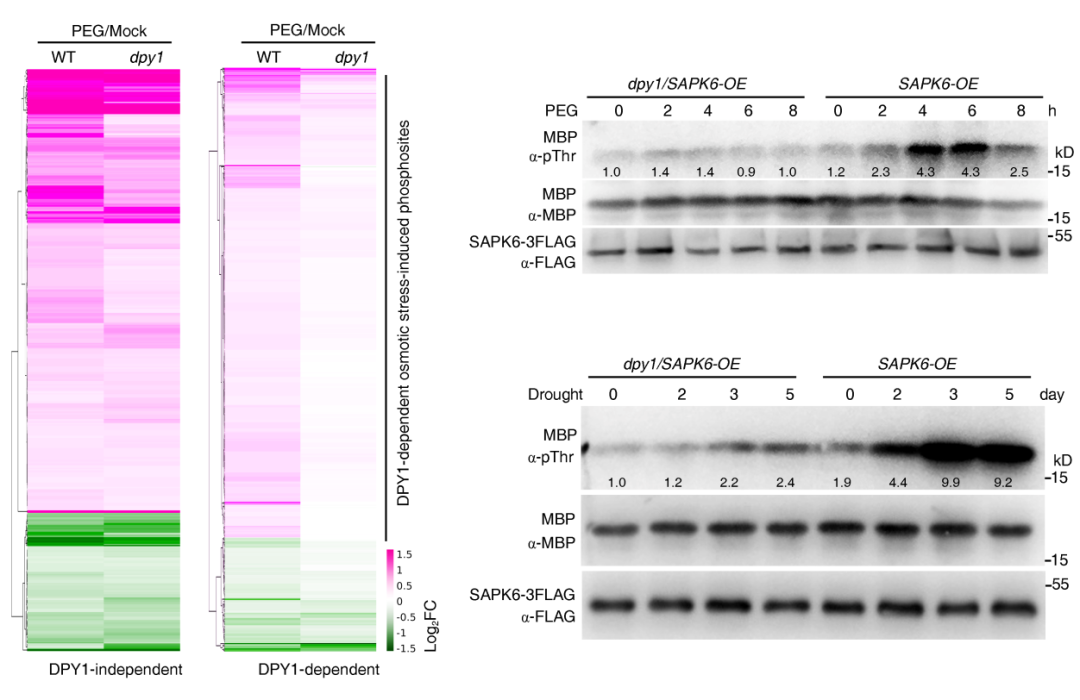
图1. DPY1控制50%以上的渗透胁迫响应磷酸化位点及SAPK6激酶的渗透激活
然而对于理解植物早期渗透胁迫信号而言,还有很多未解之谜等待进一步澄清:细胞膜上的DPY1是否直接感受胁迫,其如何被渗透胁迫激活?DPY1如何将渗透胁迫信号传递给下游的SnRK2,连接它们的蛋白有哪些?
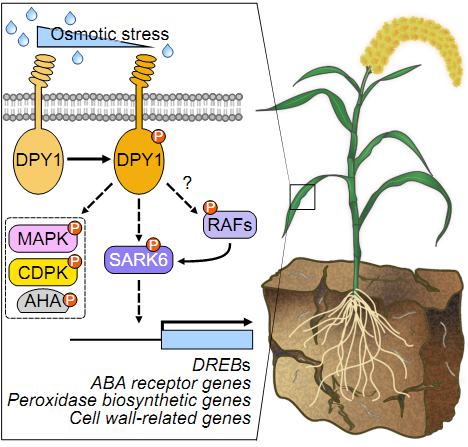
图2. DPY1介导的干旱信号转导模式图
参考文献:
1. Zhao, M., Tang, S., Zhang, H., He, M., Liu, J., Zhi, H., Sui, Y., Liu, X., Jia, G., Zhao, Z., Yan, J., Zhang, B., Zhou, Y., Chu, J., Wang, X., Zhao, B., Tang, W., Li, J., Wu, C., Liu, X., and Diao, X. (2020). DROOPY LEAF1 controls leaf architecture by orchestrating early brassinosteroid signaling. Proceedings of the National Academy of Sciences 117, 21766-21774.
2. Tang, S., Zhao, Z., Liu, X., Sui, Y., Zhang, D., Zhi, H., Gao, Y., Zhang, H., Zhang, L., Wang, Y., Zhao, M., Li, D., Wang, K., He, Q., Zhang, R., Zhang, W., Jia, G., Tang, W., Ye, X., Wu, C., and Diao, X. (2023). An E2-E3 pair contributes to seed size control in grain crops. Nature Communications 14, 3091.
3. He, Q., Tang, S., Zhi, H., Chen, J., Zhang, J., Liang, H., Alam, O., Li, H., Zhang, H., Xing, L., Li, X., Zhang,W., Wang, H., Shi, J., Du, H., Wu, H., Wang, L., Yang, P., Xing, L., Yan, H., Song, Z., Liu, J., Wang, H., Tian, X., Qiao, Z., Feng, G., Guo, R., Zhu, W., Ren, Y., Hao, H., Li, M., Zhang, A., Guo, E., Yan, F., Li, Q., Liu, Y., Tian, B., Zhao, X., Jia, R., Feng, B., Zhang, J., Wei, J., Lai, J., Jia, G., Purugganan, M., and Diao, X. (2023). A graph-based genome and pan-genome variation of the model plant Setaria. Nature genetics 55, 1232-1242.